BEEM – Biologie, Evolution et Ecologie Moléculaires
Présentation
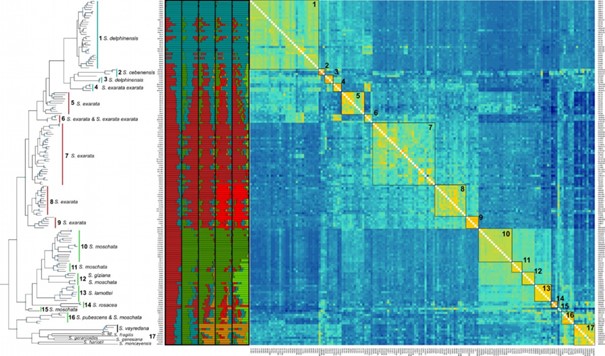
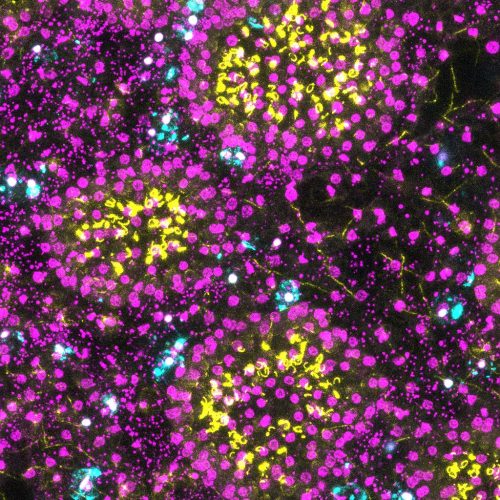
Depuis 2012, notre équipe s’est construite autour des moyens d’investigation de l’ADN pour étudier la diversité, l’évolution et l’écologie des organismes. Nos modèles d’études sont des organismes appartenant aux métazoa, viridiplantae, rhodophyta, et des communautés de micro-organismes de très nombreux phylums, en milieu marin, aquatique ou émergé. Nos thèmes de recherche sont ceux qu’on peut aborder grâce au socle conceptuel fort de la biologie évolutive et aux méthodes universelles d’étude des génomes : la systématique, l’évolution biologique, la génétique, l’écologie des communautés et la réponse éco-évolutive des organismes à un changement environnemental. Certains de nos membres sont plus biologistes et ont des approches expérimentales, d’autres sont plus des analystes et des modélisateurs de la diversité, mais nous partageons tous un émerveillement pour la fantastique histoire du vivant, sa complexité et ses rebondissements.

Thèmes de recherche
L'Équipe
Collaborations
- University of Alberta (Canada)
- Queensland University (Australia) – Partenaire Projet ABISS « INTERNATIONAL CALL 2024 “MARINE BIOLOGY”-CNRS) (E. Renard (BEEM).
- Vanuatu Fisheries Departement (VFD)
- Albion College, Michigan, USA
- University of Alabama, USA
- University of Bolgna (Italia)
- Swiss Federal Institute of Aquatic Science and Technology, Dübendorf, Switzerland
- University of Oran (Algeria)
- Molecular Taxonomy Laboratory, Hungarian Natural History Museum, Budapest, Hungary
- Ogata Lab, Université de Kyoto Japon
- Laboratoire Chimie de l’Environnement (LCE).
- Centre Europeen de Recherche et d’Enseignement de Geosciences de l’Environnement (CEREGE).
- Institut d’Océanographie Marine (MIO).
- Institut de Biologie du Développement de Marseille (IBDM). Partenaire ANR EvolAj “Structure and functions of intercellular epithelial junctions: an evolutionary perspective” (2021-2025, C. Borchiellini (BEEM)).
- Institut de Biologie de l’Ecole Normale Superieure (IBENS). Partenaire ANR OriSigne “Aneural origin of ionotropic glutamate receptor signalling”(2022-2026, E. Renard (BEEM)).
- Agence Nationale de Sécurité sanitaire, alimentation, Environnement et travail (ANSES).
- UMR Écologie Marine Tropicale des Océans Pacifique et Indien (UMR ENTROPIE).
- Institut Français Recherche Exploitation de la MER (IFREMER)
- Station de recherches sous-marines et océanographiques de Calvi (STARESO)
- Stella Mare Université de Corse
- Université du Mans
- Station marine de Concarneau
- Université de Montpellier (MARBEC)
- Observatoire Océanologique de Banyuls-sur-Mer (LECOB)
- Institut de la mer de Villefranche
- Institut Français de Bioinformatique
- DASS (Direction des Affaires Sanitaires et Sociales), Nouméa, Nouvelle-Calédonie
- Province des Iles Loyauté, Nouvelle-Calédonie
- SSPH (Service de la Santé Publique et des Handicapés) de la ville de Marseille